A gombák családfái segíthetnek választ adni evolúciónk ősi kérdéseire
Nagy László és szegedi kutatócsoportja a gombák evolúciós történetének feltárásától indult, és most, az Európai Kutatási Tanács támogatásával soksejtű létünk legalapvetőbb kérdéseire keresi a választ. Genomikai forradalom, evolúciós családfakutatás, lüktető géncsaládok és gombák minden mennyiségben – részletes összefoglalónk a kutatásról.
Ritkán fordul elő, hogy egy innovációs versenyt lefújnak, mert a kiötlésétől a kiírásáig eltelt pár évben egyszerűen túllép rajta a technológiai fejlődés. Márpedig a tízmillió dolláros Archon Genomics X PRIZE-zal pontosan ez történt. 2006-ban még őrületes vállalkozásnak tűnt egy teljes humán genom szekvenálása 30 napon belül mindössze 1000 dollárért, hét évvel később, a pályázat meghirdetésekor viszont a piacon elérhető módszerek is kezdték megközelíteni ezt a szintet. A genomika tudománya nagykorúvá vált.
Egyetlen évtized sem kellett hozzá, hogy egyes gének hatásának aprólékos tanulmányozása helyett hirtelen korábban elképzelhetetlen adatmennyiség szakadjon a kutatók nyakába. Talán azzal a hasonlattal lehetne érzékeltetni a változást, mintha eddig katalóguscédulák hosszas böngészése után egy kedves könyvtáros kiadta volna a keresett könyvet, most pedig hirtelen a kezünkbe nyomnák az egész könyvtár összes könyvének tartalmát egy pendrive-on.
De mit kezdjünk ezzel az elképesztő mennyiségű adattal?
Attól még, hogy minden ott van a gépünkön, nem fogunk gyorsabban olvasni – vagyis megfejteni az egyes gének konkrét, szervezeten belüli szerepét. A genomikával foglalkozók nem is igazán „olvasni” szeretnének, hanem inkább kihasználni ezt a hatalmas adatmennyiséget és az összefüggésekből, statisztikákból hasznos felismerésekre jutni.
Egy könyvtáros például valószínűleg üveges tekintettel nézne ránk, ha feltennénk neki azt a kérdést, hogy az ezredforduló után megjelent könyvek közül melyikben fordul elő egyszerre Arany János és Réz András neve. Az efféle feladatok azonban könnyedén megoldhatók, ha a zsebünkben lapul az a bizonyos pendrive.
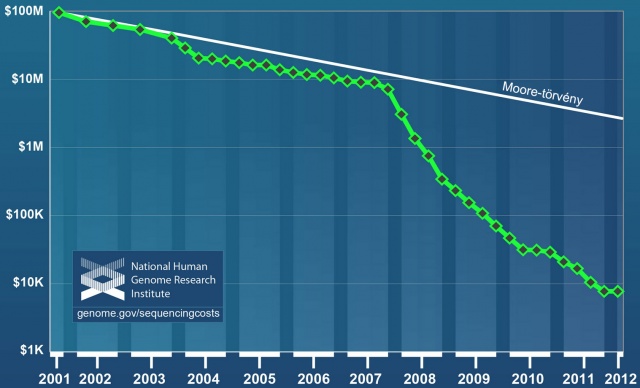 Egy teljes emberi genom szekvenálásának költsége logaritmikus skálán. A fehér egyenes a számítástechnikai eszközök költségcsökkenését jól közelítő Moore-törvény diktálta ütemet mutatja. A 2007 utáni döbbenetes letörés az új generációs szekvenátorok megjelenésének köszönhető. Forrás: National Human Genome Research Institute
Egy teljes emberi genom szekvenálásának költsége logaritmikus skálán. A fehér egyenes a számítástechnikai eszközök költségcsökkenését jól közelítő Moore-törvény diktálta ütemet mutatja. A 2007 utáni döbbenetes letörés az új generációs szekvenátorok megjelenésének köszönhető. Forrás: National Human Genome Research InstituteÚjragondolt rokonság
Persze a genomikában ennél jóval értelmesebb kérdések is feltehetők, és hamar nyilvánvalóvá vált, hogy a rengeteg adat közti kapcsolatok segíthetnek maguknak az adatoknak az értelmezésében is. A DNS-kód amellett, hogy tervrajzként tartalmazza az élőlények felépítését, egyúttal az evolúció történetének pillanatfelvétele is. Így, ha több élőlény genomját tudjuk egyszerre vizsgálni, az adatokból felállítható egy evolúciós családfa, amely megmutatja, hogy az összehasonlított élőlények őseinek fejlődése mikor vált el egymástól –vagy másképpen szólva: milyen közeli rokonsági kapcsolatban állnak egymással.
Ilyen családfákat közelebbi rokon fajok esetében akár néhány rövidebb DNS-szakasz vizsgálatával is el lehet készíteni. Így eshetett meg, hogy az ezredforduló környékén a valós, genetikai rokonságok feltárásával igencsak felbolydult a rendszertan: látszólag közeli rokon fajokról kiderült, hogy csak „unokatestvérek”, míg az is előfordult, hogy két távolabbi rokonnak tartott fajt szerencsésebb lenne „testvérnek” nevezni. Ezért, aki a kilencvenes években tanult biológiát, ma sok fajcsoport esetén teljesen új latin nevekbe futhat bele (kedvenc példám a cinkék ízekre szedett Paridae családja).
 Széncinege és kékcinege egy téli etetőn. Korábban egy nemzetségbe sorolták őket, de mitokondriális DNS-ük egy szakasza elárulta, hogy a rokonság távolabbi, mint gondolták. Forrás: stockfresh.com
Széncinege és kékcinege egy téli etetőn. Korábban egy nemzetségbe sorolták őket, de mitokondriális DNS-ük egy szakasza elárulta, hogy a rokonság távolabbi, mint gondolták. Forrás: stockfresh.comAz evolúció ugródeszkája
De miként változik a genom az evolúció során, hogyan távolodnak el egymástól a családfa ágai? Sok esetben korábbi, rendszerint már működő gének apró módosulásai (mutációi) hozhatnak apránként kicsiny változásokat, és azok a változatok tudnak fennmaradni, amelyek legalábbis nem rontják az őket hordozó élőlény túlélési esélyeit. Ezt a mechanizmust nagyjából minden középiskolás biológiakönyv tárgyalja, azonban van vele egy aprócska gond: az élet nem ennyire egyszerű.
Ha ugyanis egy gén egy jól működő funkciót kódol, akkor az élőlénynek nagy szüksége van rá. Vagyis olyan mutációsorozatot várunk el, mely egyetlen lépésben sem rontja az eredeti funkciót, és végül apró lépések sorozatán keresztül eljut az új funkcióhoz. Nos, ha ez lenne az evolúció egyetlen eszköze, valószínűleg még valahol a vízben úszkálnánk, és épp a soron következő osztódásra várnánk. Szerencsére azonban a DNS nemcsak pontonként hibásodhat meg, hanem a sejtosztódás során egész szakaszok duplázódhatnak meg vagy tűnhetnek el, így a génekből fölös példányok jöhetnek létre. Noha nagy változásnak tűnhet, e génduplikáció közvetlen hatása gyakran elhanyagolható.
A gének másodpéldányainak megjelenése viszont hihetetlenül hatékony genetikai ugródeszka, hiszen így az eredeti gén funkciója akkor is megmarad, ha az egyik példánya változni kezd. A génduplikáció lényegében szabad kísérletezési lehetőséget ad az evolúciónak – sokan az „evolúció nyersanyagaként” is emlegetik –, így nem véletlen, hogy ha az élőlények új kihívással találják szembe magukat, arra gyakorta a gének példányainak megszaporodása és a génváltozatok kialakulása lesz a válasz. Ha pedig a gén által kódolt funkció feleslegessé válik, a felhalmozódó mutációk és a duplikációhoz hasonlóan bekövetkező génkiesések hamar eltüntetik a fölös példányokat.
A génduplikációt sokan az evolúció legfőbb hajtóerejének tartják, és annyit már biztosan tudunk, hogy az élőlények genetikai múltjában jelentős szerepet kap a növekedő és összehúzódó géncsaládok lüktetése.
Nyomozás a családfán
Mindez felvet egy izgalmas lehetőséget: ha egy élőlény megoldást talál egy környezeti problémára, egy biokémiai kihívásra, röviden, ha megjelenik egy új életfunkció, az sok esetben géncsaládok növekedésével jár együtt. Ha pedig ez a funkció lényegtelenné válik, illetve eltűnik, a hozzá tartozó géncsaládok is összezsugorodnak. Érdemes azt is tudnunk, hogy a megoldásokat megvalósító funkciók mögött a legtöbbször bonyolult, nehezen felderíthető biokémiai mechanizmusok állnak, melyek kialakításában több (gyakran több tucat vagy akár több száz) géncsalád vesz részt.
Ha a genom rengeteg evolúciós változása közül egy ügyes módszerrel ki lehetne szűrni, hogy melyek azok a géncsaládok, amelyek egy ilyen bonyolult funkció megjelenésével és eltűnésével összhangban növekednek és húzódnak össze, abból következtetni lehet a hozzájuk tartozó fehérjékre, azokból pedig a biokémiai folyamatokra.
Vagyis beteljesül a genomika egyik nagy ígérete: pusztán a genetikai adatokból és a megfigyelt jelenségekből megalapozott biokémiai hipotézisek születhetnek.
Nagyjából ezt kínálja a COMPARE nevű statisztikai módszer, melyet az MTA Szegedi Biológiai Kutatóközpont Lendület-csoportvezetője, Nagy László és kutatótársai fejlesztettek ki. A szegedi biológusok gombák evolúcióját vizsgálják, eredményeik azonban, amint később látjuk majd, jóval túlmutatnak ezen az élőlénycsoporton.
 Nagy László (jobb szélen) és szegedi kutatócsoportja (a képről hiányzik Varga Torda) Fotó: mta.hu/Gilicze Bálint
Nagy László (jobb szélen) és szegedi kutatócsoportja (a képről hiányzik Varga Torda) Fotó: mta.hu/Gilicze BálintGéncsaládok szinkrontánca
A statisztikai módszer használatához mindenekelőtt több faj teljes genomjára van szükség. Ezekből a genomokból matematikai módszerekkel felépíthető egy evolúciós családfa, melyben az elágazások a fejlődési irányok szétválásait jelzik. Az így elkészült családfában nyomon követhető, hogy a vizsgált élőlények különféle funkciói (vagyis a korábban említett összetett biokémiai megoldások) melyik elágazásnál jelentek meg, illetve tűntek el. Ugyanezen a családfán az is látható, hogy elágazásról elágazásra haladva mely géncsaládok növekedtek, illetve húzódtak össze.
Most pedig már csak az van hátra, hogy a kiválasztott funkció megjelenésének és eltűnésének mintázatát összevessük a géncsaládok lüktetésének térképével – ebből a COMPARE képes kimutatni, hogy mely géncsaládok érintettek nagy valószínűséggel a funkció megvalósításában. Fontos azonban a módszer használhatóságához, hogy ne csak közeli rokon fajok genomját vizsgálják, mert itt sok géncsalád lüktetése a funkció evolúciós történetétől függetlenül is hasonló, egyszerűen a szoros rokoni kapcsolat miatt. Ezért van szükség viszonylag sok, ráadásul különféle rokonsági kapcsolatban álló élőlény genomjára.
Szerencsére ezeket tálcán kínálja a nemzetközi együttműködésben zajló 1000 gombagenom projekt, melynek célja, hogy lefedje a gombák birodalmának teljes genomkönyvtárát, mintát véve az összes jelentősebb fajcsoportból. A szegedi kutatók bekapcsolódtak a projektbe, és az amerikai Joint Genome Institute-tal közösen olyan gombafajok genomját tárják fel, melyek saját kutatási irányaikat is segíthetik.
Akinek a fenti rövid leírás kicsit elvontak tűnik, és szeretné némileg képszerűbben látni a módszert, annak szól a következő keretes írás – a többiek nyugodtan görgessenek lejjebb.
Sorozatfüggők a betondzsungelben
Képzeljük el, hogy egy közvélemény-kutató cég megbízott azzal, hogy derítsük fel a méltán népszerű Érzelmek közt című tévésorozat megszállott rajongóit. Módszerünk egyszerű, jóllehet kissé személytelen: egy panelház ablakait bámulhatjuk esténként. Az ablakokban felbukkanó ismerős kékes fény jelzi, hogy egy család leült a tévé elé, ha pedig a kékes fény eltűnik, kikapcsolták a tévét. (Talán nem túl merész asszociáció, ha az ablakok a géncsaládoknak felelnek meg, a kék fény felvillanása és eltűnése egy-egy géncsalád növekedésének és összehúzódásának, egy végigkövetett este pedig egy élőlény rekonstruált evolúciójának leképezése egészen a családfa gyökerétől kiindulva.)
 Forrás: Flickr/Vladimir Varfolomeev
Forrás: Flickr/Vladimir VarfolomeevA sorozat valódi rajongói azok a (gén)családok, amelyek pontosan akkor kapcsolják be a televíziót, amikor elkezdődik egy Érzelmek közt-rész, ha pedig befejeződik, ki is kapcsolják a készüléket.
Világos, hogy ha csak egy napon vagy pár egymást követő napon (lásd: közeli rokonok) figyeljük az ablakokat, abból nem tudhatunk meg túl sokat, hiszen könnyen lehet, hogy például egy másik csatorna híradórajongói vagy egy természetfilm lelkes nézői is éppen abban az időpontban kattannak rá a távirányítóra.
Viszont rengeteg napot megnézhetünk (sok faj genomját használhatjuk), ráadásul ezeken a napokon változó lehet a többi csatorna kínálata, más és más időpontban megy adásba az Érzelmek közt, sőt lehet dupla epizód, esetleg Érzelmek közt-maraton, vagy akár el is maradhat az adás (a funkció távoli rokonoknál máshol, más környezetben jelenik meg és tűnik el), ugyanakkor a tévéműsort pontosan ismerjük (a családfából kikövetkeztethető a funkció története).
Akármelyik napot nézzük is, egyet biztosan tudunk: a valódi rajongók ablakainak kékes villódzásai mindig kedvenc sorozatuk adásával lesznek szinkronban – akárhova tegyék is a műsorban, és akárhány részt vetítsenek is. Ha pedig elég sok és kellően különböző napot követünk, szépen előtűnnek a rajongók szobái a panelház villogó ablakainak háttérzajából.
Ezt az intuitív képet, ezt az „előtűnést” formalizálja a szegedi kutatók statisztikai módszere.
A gombák ismeretlen fegyverei
A nagy reményű módszer első igazi próbája során Nagy László és kutatótársai annak eredtek nyomába, hogy mi állhat az ún. fehérkorhasztó gombák működésének hátterében. Ezek a gombák olyan enzimkészlettel rendelkeznek, mely alkalmassá teszi őket nemcsak a cellulóz, de a fás szárú növények sejtfalának sokkal ellenállóbb komponense, a lignin lebontására is.
Egy korábbi kutatásból kiderült, hogy a ligninbontás képessége ősi evolúciós vívmány, mely nagyjából 300 millió évvel ezelőtt jelenhetett meg a mai kalapos gombák őseiben (egészen pontosan az Agaricomycetes osztályban). Az innováció jelentőségét és biokémiai összetettségét jól jelzi, hogy ez a képesség sehol máshol nem bukkant fel a földi élet történetében. Egy elmélet szerint az sem véletlen, hogy épp ekkor ért véget a földtörténeti karbon kor: a fehérkorhasztó gombák immár képesek voltak a ligninben tárolt szenet visszajuttatni a szénkörforgásba – ezzel lezárult a kőszénlerakatok felhalmozódásának időszaka.
A ligninbontás kémiai eszköztárának fenntartása azonban energiaigényes feladat, hiszen rengeteg enzimet kell a gomba sejtjeinek környezetébe juttatni. Ezért lehetnek olyan helyzetek, amikor nem ez a legalkalmasabb módszer a tápanyagok felvételére – ilyenkor e nagyon hatékony enzimekhez tartozó géncsaládok háttérbe szorulnak.
Nagy László és kutatótársai vizsgálatukban 62 gombafaj teljes genomját vetették össze a COMPARE módszerrel. A gombafajok között voltak fehérkorhasztók, a lignint bontani nem vagy csak módosítani képes barnakorhasztók, valamint a növényekkel szoros kapcsolatban élő ektomikorrhizás gombák (ez utóbbi csoportba tartoznak például a szarvasgombák vagy a mesekönyvekből is ismert légyölő galóca – ami szintén szerepelt a vizsgálatban).
Az eredmények igazolták a módszerrel kapcsolatos legvérmesebb reményeket is: amellett, hogy nagyrészt azonosították azokat a géncsaládokat, amelyekről eleve tudták, hogy részt vesznek a fehérkorhasztó gombák működésében, több tucatnyi új géncsaládra is rábukkantak, melyekről az élettannal foglalkozók nem is sejtették, hogy fontos résztvevői ezeknek a folyamatoknak, azonban korábbi, a gének kifejeződését leíró adatok vizsgálatával sikerült a szerepüket igazolni.
Látványos volt az is, hogy az ektomikorrhizás gombákban mennyire visszaszorultak a ligninbontás enzimjeit kódoló gének. A kutatók számítottak is erre, hiszen itt a gomba és a növény gyökere között finoman hangolt kapcsolat áll fenn, és ebben a rendszerben minden bizonnyal túl nagy pusztítást okoznának ezek a hatékony sejtfalbontó enzimek.
Hogyan mérjünk evolúciós időt?
Az evolúciós családfák vizsgálata legalább annyira régészet, mint biológia. Az élőlények DNS-kódjának egyes szakaszai „molekuláris óraként” használhatók, mivel bennük átlagosan egyenletes ütemben jelennek meg újabb és újabb mutációk (már ha generációkat számolunk). Ez a molekuláris óra azonban nem minden szakaszon jár ugyanolyan sebességgel – egy fontos enzimet kódoló DNS-szakasz például lassabban változik, mint egy nem kódoló szakasz. Ennek egyszerűen az az oka, hogy a fontos enzimet kódoló szakaszon megjelenő mutációk nagy része káros, tehát rövid úton eltűnik az élőlény kiszelektálódásával.
Amikor több élőlény genomjának összehasonlításával evolúciós családfát készítenek a kutatók, az efféle molekuláris órák segítségével képet kaphatnak a családfa csomópontjainak viszonylagos időbeli távolságáról, ezek az adatok azonban nem fordíthatóak le egyszerűen évmilliókra. Egyrészt hosszú, több százmillió éves időtartamon keresztül nem használhatók ugyanazok a molekuláris órák, másrészt hosszú távon az órák sebessége is módosulhat a DNS-szakasz szerepének és az élőlény életciklusának változása miatt is. Ahhoz, hogy ezeket az eltéréseket összeegyeztessék, és a családfa csomópontjaira valódi évszámokat írjanak, szükség van valamiféle kalibrációra, amiben független forrásból szerzett fejlődéstörténeti adatok segíthetnek.
A legtöbb mai gombafaj például helyhez kötött, spóráit pedig egyszerűen a környezetére – szélre, vízre, állatokra – bízza. Azonban akadnak olyan fajok is, amelyek egyik életszakaszukban ostorral képesek magukat hajtani a vízben. A különféle gombafajok genomjának összehasonlításával kapott családfából látszik, hogy az ostor kialakításához kapcsolódó gének ott lehettek az ősi gombákban, majd egy viszonylag gyors átmenet során sokan hirtelen elvesztették e gének többségét: ez volt az az időszak, amikor a gombák meghódították a szárazföldet.
Ha jól ismert kapcsolat – például szimbiózis – áll fenn egyes gombák és más élőlények között, e partnerek ismert korú fosszíliái teremthetik meg az időbeli kalibráció lehetőségét. Különösen nagy segítséget adhatnak azok a gombafajok, amelyek növényekkel alakítanak ki valamilyen szoros viszonyt. Az az időszak, amikor fejlődésük során meghatározóvá váltak a gomba-növény kapcsolathoz szükséges géncsaládok, feltehetőleg egybeesik a partnernövény elterjedésének idejével, erről pedig gyakran a fosszíliák árulkodnak.
Azt, hogy a módszer kevésbé ismert terepen is jól működik, a szegedi kutatók egy nemrég megjelent cikkükben bizonyították. Ezúttal azt mutatták ki, hogy a tuskógombák gyökérszerű nyúlványainak, a rizomorfoknak a növekedésében ugyanazok az ősi géncsaládok játszanak fontos szerepet, amelyek a termőtest (vagyis a közismert kalapos gomba) kialakításában is. Ez az eredmény pedig már elvezet ahhoz, ami a következő öt évben vár Nagy Lászlóra és csapatára – az Európai Kutatási Tanács (ERC) nyertes pályázatának megvalósításához.
 Tuskógomba termőtestei és a fekete, cipőfűzőszerű rizomorfok – Nagy Lászlóék kimutatták, hogy felépítésüket ugyanaz az ősi genetikai eszköztár segíti Forrás: Wikimedia Commons/Jerzy Opioła – CC-BY-SA
Tuskógomba termőtestei és a fekete, cipőfűzőszerű rizomorfok – Nagy Lászlóék kimutatták, hogy felépítésüket ugyanaz az ősi genetikai eszköztár segíti Forrás: Wikimedia Commons/Jerzy Opioła – CC-BY-SAVan-e élet a génduplikáción túl?
A gombák életüket apró spóraként kezdik. Ha a körülmények kedvezőek, a spórából gombafonalak kezdenek növekedni, majd ezek újabb és újabb elágazások révén bonyolult szövedékként behálózzák a talajt vagy éppen a zacskóban felejtett, múlt heti párizsis szendvicset. A gombafonalakon belül mindössze néhány sejttípus létezik, és a gombafonalakból álló szövedék alapvetően „demokratikus” – nincsenek olyan jól elkülönülő és egészen különböző szerepet ellátó szövetek, mint mondjuk, a fejlettebb növényeknél. Azonban számos gomba életében elkövetkezik egy pillanat, amikor a gombafonalak kuszaságában megjelenik egy kicsiny csomó, és ebből fejlődni kezd a termőtest – így születik meg a mindenki által ismert csiperke, a gyilkos galóca vagy a nagy őzlábgomba.
Ezekben a termőtestekben viszont egészen eltérő szerepet kapnak a sejtek, tehát a gomba növekedése során valahonnan „tudniuk” kell, mi lesz a feladatuk. Itt már sok sejttípus van, ilyen szempontból a termőtestek hasonlítanak a fejlett növényekre vagy állatokra. Ezt nevezik a biológusok komplex soksejtűségnek szemben az gombafonalak kusza hálózatánál tapasztalható egyszerű soksejtűséggel. Így hát egyetlen gomba élete során mintha több százmillió éves evolúciós folyamaton száguldanánk végig – és ennek a folyamatnak a forgatókönyve ott kell hogy legyen a gombák DNS-ében.
 Fejlődésnek induló apró termőtestek a szegedi kutatók laborjában Fotó: mta.hu/Gilicze Bálint
Fejlődésnek induló apró termőtestek a szegedi kutatók laborjában Fotó: mta.hu/Gilicze BálintDe vajon hol rejtőzhet ez a forgatókönyv?
Némi leegyszerűsítéssel úgy érdemes elképzelni, mintha a kifejlett élőlények összerakott legójátékok lennének — a fehérjéket kódoló gének a különféle építőkockák, a gének közti szakaszokban pedig valahol ott rejtőzik az összerakás tervrajza. Jóllehet a géncsaládok növekedése és összeesése nagyszerűen rámutat számos biokémiai trükkre, vagyis magukra a legókockákra, a kutatók mégis biztosak benne, hogy a tervrajznak vannak további, rejtett részletei.
Egyik tervük, hogy összehasonlító módszerüket úgy szabják át, hogy érzékennyé váljon a gének kifejeződését szabályozó DNS-részletek evolúciós változásaira. Vagyis abban bíznak, hogy sikerül valamiképp ráakadni azokra a pontokra, ahol a tervrajz lényeges változásokon megy át az evolúció során. Mivel ezek a változások a komplex soksejtűség alapjait jelentik, elképzelhető, hogy ismeretük a jelenség általános megértésében is segít majd. Az ismeretlen géncsaládokhoz hasonlóan itt is felbukkanhatnak olyan, eddig nem is sejtett jellemzők, melyeket azután másutt is meg lehet keresni. Ebben – és más genomikai vizsgálatokban is – nagy segítséget jelent, hogy a gombák DNS-kódja kompakt, vagyis igen kevés „fölös” információ van a konkrét fehérjéket kódoló génjeik között (ellentétben például az emberrel, ahol a gének összességében néhány százaléknyi „oázisként” jelennek meg a fehérjét nem kódoló DNS-szakaszok sivatagában). Ez is lehet az oka, hogy az első ismert teljes genomok között jelentős számban vannak gombák DNS-kódjai.
Ezekben a vizsgálatokban is sokat segíthet a korábban már említett 1000 gombagenom projekt, mely ha lezárul, a gombák birodalmát lefedő 500 család mindegyikéből 2-2 faj teljes genomját teszi elérhetővé a kutatók számára. Hogy mikor, az most már leginkább tudományos érdeklődés kérdése, hiszen a szegedi kutatók partnere, az amerikai Joint Genome Institute egy beküldött mintából 3 hónap alatt kiolvassa a teljes genomszekvenciát, és statisztikai vizsgálatokra alkalmas adathalmazt állít össze belőle.
Az elnyomás alternatívái
A szegedi biológusok másik érdekes kutatási iránya egészen más szinten vizsgálja a komplex soksejtűség eredetét. Ahhoz, hogy a DNS-kódból konkrét fehérjék keletkezzenek, a génekben tárolt információknak el kell jutniuk a sejtek „fehérjegyáraihoz”, vagyis a riboszómákhoz. Az információ hordozója a hírvivő RNS, mely némi leegyszerűsítéssel olyan, mint a DNS-molekula egyik szálának egy szakasza: a genetikai kód betűi sorakoznak rajta egymás után.
Amikor azonban egy gén DNS-kódjából a sejtmag átírómechanizmusa hírvivő RNS-t készít, az nem minden esetben a végleges, fehérjét leíró kódsorozat. A sejtmag enzimjei ugyanis sok esetben kivágnak belőle egyes részeket, és ez a végtermék juthat el a riboszómákhoz. Egy DNS-kódból tehát többféle hírvivő RNS, így többféle fehérje jöhet létre: ez az alternatív splicing.
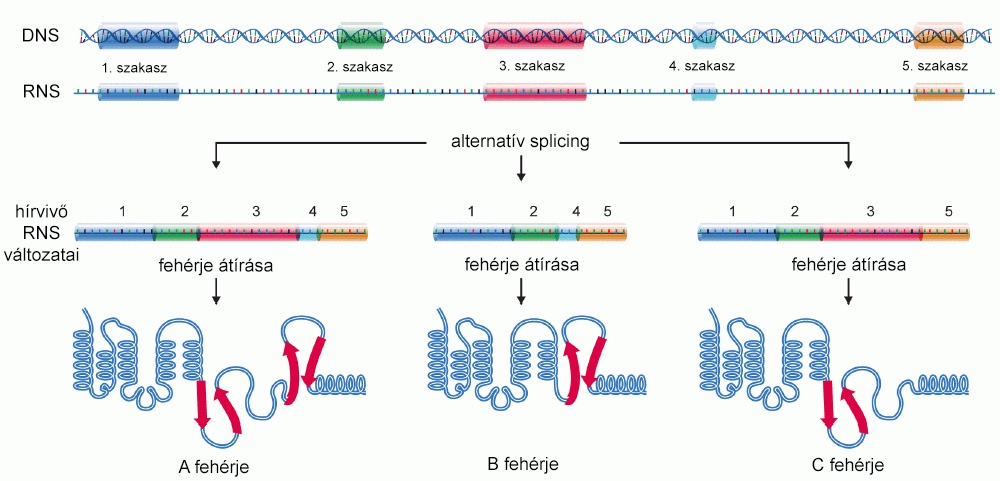 Az alternatív splicing mechanizmusának vázlata Forrás: Wikimedia Commons, mta.hu
Az alternatív splicing mechanizmusának vázlata Forrás: Wikimedia Commons, mta.huÉrdekes módon még e különféle módokon összetapasztott hírvivő RNS-ek sem mind testesülnek meg valódi fehérjékben: bizonyos, sejtben működő mechanizmusok kiszűrik egy részüket, mielőtt eljutnának a riboszómákhoz.
Elképzelhető, hogy az alternatív splicing és e szűrőmechanizmus segítségével a sejt környezetétől vagy az időtől függően szabályozni tudja, hogy adott génből melyik fehérjeváltozat keletkezzen és milyen koncentrációban. Ezért hát kiváló módszernek tűnik arra, hogy a sejtek életük során, illetve környezetük változásával más és más szerepet töltsenek be – éppen erre van szükség a komplex soksejtűség kialakulásához.
A szegedi kutatók arra kíváncsiak, hogy miként járul hozzá az alternatív splicing a sejttípusok sokféleségének kialakulásához. Fontos kérdés, hogy a rengeteg hírvivő RNS-változat közül melyekből készülnek valóban fehérjék, és melyek azok, amelyek esetleg csak azért vannak épp jelen, hogy a megfelelő időben elnyomják az adott gén kifejeződését.
Magukat a fehérjéket költséges és bonyolult vizsgálni, és gyakran a hírvivő RNS-ek kódja sem árulja el, hogy végül nem lesz belőlük fehérje. Nagy László és kutatótársai így ismét a genetikai családfákhoz fordultak. A következőkben azt vizsgálják majd, hogy a rengeteg hírvivő RNS-változat közül melyek változtak gyorsabban, és melyek lassabban az evolúció során. Ezekből a különbségekből reményeik szerint következtetni lehet majd az egyes változatok szerepére, ami sokat elárulhat az alternatív splicing mechanizmusáról – ez pedig jóval túlmutat a gombák világán.
Az ERC-nyertes kutató és csapata gombák genetikai kódjával foglalkozik, azonban a kutatások eredményei jó eséllyel általános tanulságokat is adnak majd, hiszen jelenlegi tudásunk szerint a komplex soksejtűség a Földön mindössze hat független esetben alakult ki – ebből kétszer a gombák körében.
További információ
Nagy László, MTA SZBK Biokémiai Intézet, Szintetikus és Rendszerbiológiai Egység, Gomba Genomika és Evolúció Csoport
+36 62 599 633








